https://link.springer.com/article/10.1007/s00330-022-09153-z
Ma, Xiaoling, et al. "Development and validation of a deep learning signature for predicting lymph node metastasis in lung adenocarcinoma: comparison with radiomics signature and clinical-semantic model." European Radiology (2022): 1-14.
Lung adenocarcinoma (LUAD) 에서, Lymph node metastasis를 예측하는 논문인데,Deep learning과 Radiomics 두가지 모델을 구성하여 machine-learning feature과 임상변수와의 관계를 비교해보는 논문이다.
메인은 Deep learning model을 만드는 것이며,Swin Transformer 라는 개념을 CNN block에 추가하여 구현하였다.
Swin Transformer
https://github.com/yingkaisha/keras-vision-transformer
GitHub - yingkaisha/keras-vision-transformer: The Tensorflow, Keras implementation of Swin-Transformer and Swin-UNET
The Tensorflow, Keras implementation of Swin-Transformer and Swin-UNET - GitHub - yingkaisha/keras-vision-transformer: The Tensorflow, Keras implementation of Swin-Transformer and Swin-UNET
github.com
2021년 Vision Transformer (ViT) 이후, Transformer가 꾸준히 이미징 처리에 인기를 얻고있다.
이 논문에서는 ViT보다 발전된 형태인 Swin-transformer를 활용하여 이미지 분류를 시도하였다.
맨 위 Architecture에서 보듯, patch 단위로 이미지를 잘라서 embedding을 수행한 이후,
Swin Transformer block를 연속적으로 활용하는데,
이 block은 Scale Swin Transformer Block, Conv, MLP + Residual connection과 함께 정보를 넘겨준다.

위 그림에서 (b) shifted window가 Shifted window (SWIN) 이라는 이름을 대표적으로 설명하는 그림이라고 볼 수 있다.
위 그림에서 회색 patch들을 Window (빨간 네모)들 안에서 self-attention을 계산하여 각 patch들에서 feature를 뽑는데,
이러한 Window 기반의 self-attention module은 서로 다른 윈도우 (빨간 네모) 간의 연결을 설명하지 못하는 단점이 있다.
이러한 단점을 극복하기 위한 SWIN 알고리즘은 Mult-head Self Attention (MSA) 를 shifted window에서 계산하게 되는데, 이게 (b)의 우측 그림이다. 따라서 그림 (c)에서, W-MSA와 SW-MSA를 거쳐 SWIN 을 통해 이미지들의 patch들에 대하여, Window간의 Over-lap 없이 매우 효율적으로 cross-window connections을 통해 이미지의 feature를 처리할 수 있다.
LN은 LayerNormalization을 의미하며, tf.keras.layers.LayerNormalization로 이미 구현된 layer이다.
MLP는 말 그대로 Multi-layer perceptron으로, 2개의 fully connected layer로 이루어져 있으며,
첫번째 layer는 gelu activation, 두번째 layer는 linear activation으로, 각 layer 다음에 dropout을 거쳐 return하게 된다.
다시 European Radiology 논문으로 넘어와, Figure 2는 LUAD에서 Lymph node meta를 예측하는 architecture이다.

CT 이미지에서, 병변이 최대가 되는 slice의 3-channel 을 input으로 활용하였다.
Radiomics 데이터는 ITK-SNAP (version 3.8.0) 를 활용하여 3D segmentation을 수행한 후,
PyRadiomics를 활용하여 feature를 추출하였다.
PyRadiomics software (https://pyradiomics.readthedocs.io/en/latest/index.html).
- First Order Statistics
- Shape-based
- Gray Level Cooccurence Matrix
- Gray Level Run Length Matrix
- Gray Level Size Zone Matrix
- Neighbouring Gray Tone Difference Matrix
- Gray Level Dependence Matrix
Radiomics feature는 조금 redundant한 편인데, 따라서 이 논문에서는 feature selection을 위하여
The intraclass correlation coefficient (ICC) 값이 0.7 이상인 feature는 제외하였다.
그리고 Lymph meta에 따른 feature 분포의 차이가 없는 변수들을 mann-Whitney test로 제외하였다. (p<0.05)
그 후, 간단한 logit 모델을 기반으로, forward stepwise selection를 활용한 regularization을 통해
lymph meta를 분류하는 모델을 학습하였다.
그리고, Clinical data 기반의 모델 또한 radiomics와 거의 비슷한 방법으로 구축하여,
최종적으로 CT Image based 딥러닝, Radiomics based Logistic model, Clinical data based Logistic Model을 비교한다.
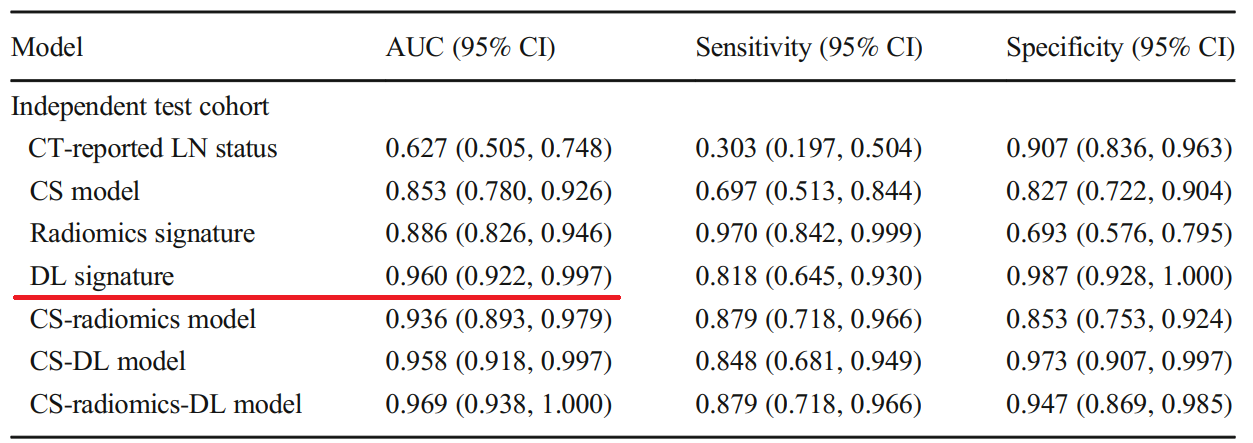
논문에서 결론은 Deep learning이 짱이라는건데,
Radiomics를 너무 기본적인 regression으로 모데링을 했다는 아쉬움이 있다.
'Major Study. > Bioinformatics' 카테고리의 다른 글
| Single Cell Analysis Best Practice 정리해보기 (1) | 2023.02.06 |
|---|---|
| GTEx에서 Pathology image 분석하기 (0) | 2022.10.13 |
| Multi-class와 Multi-label classificiation, 분포가 다른 Output들 (0) | 2022.08.18 |
| deep learning for the life sciences (3) | 2022.06.12 |
| Bioinformatics 공부용 웹사이트 rosalind.info (0) | 2021.08.04 |